Sommaire
- Vaccines & Endotoxin, a Challenging World.
- Méthode alternative d’identification. Exemple d’une implémentation d’un système Maldi Tof au laboratoire de contrôle.
- Implantation d’une méthode microbiologique alternative et polyvalente au laboratoire. Exemple du BACTALERT 3D®
- From Variable Operator Numeration To The Standardized 3P® Station Automated Colony Counting On Environmental Monitoring Culture Media Plate
- Analyse de la charge microbienne en temps réel pour prévenir les risques de contamination et mieux contrôler les procédés.
- ISO 22519 PW/WFI production systems standard: Quality aspects.
- Eau pour préparations injectables en vrac (productions, coûts). Étude de cas axée sur la qualité microbiologique.
- Coming to basics on rouging.
- Winning combination: Reducing particles in RTU packaging by aligning lid material, adhesive and sealing parameters in a holistic approach.
Méthode alternative d’identification. Exemple d’une implémentation d’un système Maldi Tof au laboratoire de contrôle.
Ces dernières années, la microbiologie pasteurienne a quelque peu été bousculée, les méthodes “rapides” ou dorénavant “alternatives” sont désormais démocratisées et même suggérées dans certains textes récents. Suivant alors la monographie Phar Eur 5.1.6 “Méthodes alternatives pour le contrôle de la qualité microbiologique”, le microbiologiste se voit alors devoir valider de la spécificité, de l’exactitude ou même étudier une robustesse. Un décryptage et un retour d’expérience sur la validation et l’implémentation réussie d’un système d’identification Maldi-Tof au laboratoire de contrôle sera discutée.
1. Introduction
Existe-t-il encore un Microbiologiste aujourd’hui n’ayant pas entendu parler des Méthodes Alternatives en Microbiologie (MAM) ? Entre les catalogues et les propositions des fournisseurs, les agendas des symposiums et des congrès, les articles de la presse spécialisée et désormais les nouvelles versions des principaux textes régissant nos quotidiens (e.g. draft Annexe 1 des BPF), il semblerait que ces méthodes soient partout. Partout oui, mais finalement très peu en routine au sein de nos laboratoires …
Excepté dans certaines grosses structures, ces méthodes et technologies sont encore peu ou pas représentées pour des analyses libératoires. Les raisons à cela peuvent être à trouver dans :
- La méconnaissance et le manque de recul sur les technologies disponibles, la difficulté de se projeter au quotidien avec une nouvelle méthode (e.g. ne plus exprimer un résultat de dénombrement en UFC),
- Les blocages internes liés aux dossiers réglementaires, changer de méthode est synonyme de variations, qui peuvent être parfois longues, coûteuses et inclus dès l’établissement du business case,
- La validation d’une méthode alternative, si la montée de version récente de la monographie Phar Eur 5.1.6 a permis une meilleure compréhension des principes et des requis alors les principes sont différents par rapport aux monographies classiques en microbiologie (i.e. 2.6.12..). Certains systèmes sont bien sûr plus simples à valider que d’autres mais désormais nous devons nous approprier le langage de nos collègues physico-chimistes, en parlant d’équivalence, d’exactitude, de spécificité, limite de quantification, de robustesse ou encore de linéarité.
- Enfin, la difficulté majeure est de justifier un investissement coûteux auprès de son organisation. Si l’exercice est plus aisé pour des cas très particuliers, comme la mise en place d’un test rapide de stérilité pour des produits finis à péremption très courte. Remplacer un test de dénombrement traditionnel peu coûteux, par un investissement de plusieurs dizaines de milliers d’euros, nécessitant des réactifs pointus et du personnel qualifié et demander des mois de travaux de validation/qualification… ceci pour gagner quelques heures ou quelques jours, peut se révéler être un combat difficile à mener.
Dans cet article est présenté un retour d’expérience sur l’implémentation au laboratoire d’un système alternatif Maldi Tof d’identification ; de la genèse du projet, au choix de la technologie, au décryptage et à la validation selon la monographie Phar Eur 5.1.6 “Méthodes alternatives pour le contrôle de la qualité microbiologique” ainsi que l’utilisation en routine.
2. Genèse du projet et choix de la technologie
Dans un souci d’amélioration continue, une évaluation sur l’optimisation des différentes activités au laboratoire a été initiée il y a quelques années. De par son volume annuel, l’identification des micro-organismes au laboratoire par un système type “biochimique automatisé” est apparue comme une activité chronophage. L’identification au laboratoire est un requis qui concerne à la fois l’analyse des produits et les monitorings environnementaux. Identifier participe à garantir la sécurité des patients, par exemple, les enquêtes objectionnables pour les produits non obligatoirement stériles, ou encore de suivre l’évolution de la flore microbienne des unités de production pour permettre de visualiser et d’anticiper les dérives. Identifier efficacement au laboratoire a donc un impact direct sur les délais de libération au laboratoire de contrôle. Les technologies d’identification alternatives disponibles à ce jour sont : la génétique (ou technologie de 3ème génération) reconnue comme “gold standard”depuis plus de 20 ans, elle est même suggérée par certains textes (i.e. FDA guideline on Sterile Drug Products, 2004). La 4ème génération : le Maldi Tof, une identification phénotypique basée sur la comparaison des profils protéolytique des cellules. Plusieurs études ces dernières années ont démontré une performance similaire à la génétique tout en étant plus rapide, moins cher et plus simple à mettre en oeuvre au quotidien au laboratoire (Chao et al., 2014).
Maldi-tof, principe pour l’identification microbienne
La colonie inconnue est directement étalée sur une plaque appelée “cible”, après ajout d’acide formique pour faciliter la lyse des cellules, une matrice “HCCA” est uniformément déposée sur l’ensemble. Son rôle est de générer des ions de différentes tailles sous l’action d’un laser. Les différents ions de m/z (masse/charge) sont alors différenciés lors de leur temps de parcours dans un champs magnétique. Les ions les plus légers arriveront plus tôt au détecteur que les plus lourds. Ce détecteur convertit alors les signaux en spectres m/z qui sont alors comparés à une bibliothèque de spectres de référence, générant alors le résultat d’identification si le score de comparaison est significatif.
3. Contexte règlementaire et évolution
Les principaux référentiels évoquant les méthodes rapides ou alternatives en microbiologie, leurs descriptions, principes de validation et d’implémentations sont :
- PDA Technical Report 33, première édition en 2000, revue en 2013
- Monographie 5.1.6 de la Pharmacopée européenne, revue en juillet 2017
- La monographie USP <1113> concerne la validation des méthodes d’identification dont les méthodes alternatives et la monographie <1123> plus largement l’ensemble des méthodes alternatives.
Pour les méthodes d’identification, les monographies dédiées Phar Eur 5.1.6 et USP <1113> sont suffisantes pour effectuer une implémentation. Si les grands principes sont proches, la vérification de méthode selon l’USP propose 3 approches différentes :
- L’analyse en parallèle de 50 isolats avec un système existant,
- L’Analyse de 12 à 15 isolats représentatifs de la variabilité taxonomique que peut identifier le système,
- Une confirmation sur 20 à 50 identifications comprenant 15 à 20 espèces différentes vis-à-vis d’un laboratoire de référence.
Concernant la monographie européenne, la possibilité que des technologies différentes puissent générer des résultats différents est clairement exprimée. Dès lors, la comparaison de systèmes de technologies différentes ou pire, de générations différentes n’est pas nécessaire. Il s’agit d’un point très important ici dans la mesure où très souvent les notions de “comparabilité” sont rapidement évoquées dès lors qu’un changement ou remplacement d’une méthode ou d’un équipement est initié. D’expérience, la réponse à donner est : “Compareriez-vous les top résultats issus de moteurs de recherche internet différents ?“. Une comparaison intéressante entre les textes Phar Eur 5.1.6 et USP <1113> a été effectuée par Tim Sandle en 2013.
4. Stratégie de validation
Si cela reste à l’appréciation de chacun, l’approche européenne nous est apparue plus cohérente dans la façon d’aborder l’implémentation d’un nouveau système. Une fois les requis de qualification de l’équipement (Qi, Qo) et du système informatique finalisés, la validation du système d’identification selon les paramètres définis dans la monographie 5.1.6 peuvent être déclinés :
- Exactitude : “Capacité à identifier le micro-organisme étudié au niveau taxonomique requis“
- Spécificité : “Capacité à opérer une discrimination entre les microorganismes effectivement présents et les facteurs d’interférence entraînant de fausses identifications”
- Robustesse : “Capacité à ne pas être affectée par des modifications faibles, délibérées, de paramètres associés à la méthode“.
La validation débute par l’étude de robustesse afin de maitriser l’ensemble du périmètre et d’estimer la totale capacité du système.
Choix des micro-organismes
Quinze micro-organismes différents ont été utilisés pour la validation (Tableau 1).
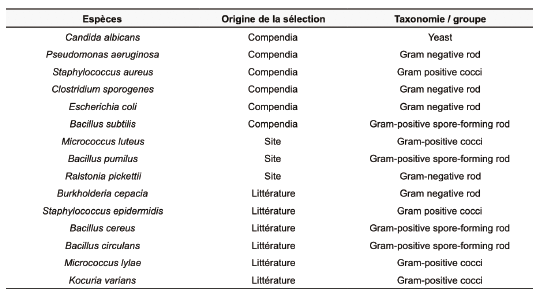
Ces micro-organismes ont été sélectionnés pour couvrir les différentes catégories taxonomiques de bactéries ou levures constituant la bibliothèque de spectres de référence.
Les paramètres de sélection ont été :
- Présence dans les principales monographies pharmacopées internationales,
- Historique de détection issu du monitoring environnemental,
- Revue de la littérature sur les tendances de contamination des environnements pharmaceutiques.
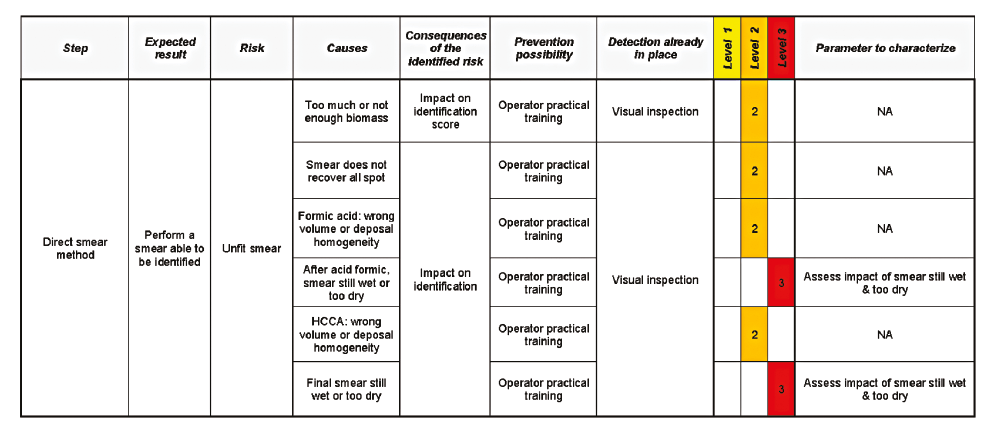
Robustesse : choix des paramètres
Les paramètres d’étude ont été définis par une approche classique “d’analyse de risque méthode”. Chaque étape de la procédure d’identification est séparée et analysée indépendamment dans sa capacité à influer sur la qualité du résultat final. Sur les 3 niveaux de criticité utilisés, seuls les paramètres identifiés comme “niveau 3” ont été soumis à étude. La procédure est divisée en : microbiologie, extraction, dépôt direct, tests, équipement, lancement analyse et réactifs. Exemple d’évaluation de l’étape “dépôt direct” (Tableau 2) : les paramètres identifiés comme nécessaires à étudier sont présentés dans le Tableau 3, avec comme principaux axes d’étude : la qualité initiale des colonies, la préparation de la cible et la qualité des réactifs.
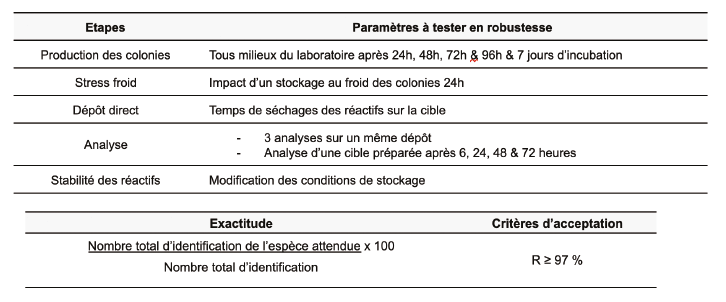
Exactitude & spécificité : modalités d’étude et critères d’acceptation
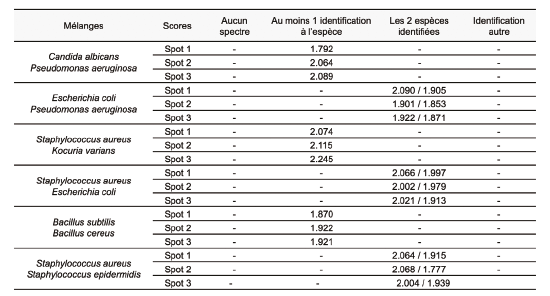
L’évaluation du critère d’exactitude est obtenue à partir des recouvrements obtenus entre le nombre d’identifications jusqu’à l’espace avec un score significatif sur le nombre total d’identifications effectuées lors de l’essai. L’identification des quinze micro-organismes a pr.alablement .t. confirm.e sur un système équivalent. Concernant le critère d’acceptation, dans la littérature la précision du système MALDI TOF pour les identifications microbiennes, se situe entre 94 et 98 % pour des micro-organismes courants selon les publications (Bilecen et al., 2015 ; Croxatto et al., 2012). Dans notre cas, un taux de précision de 97% a été choisi, cette valeur correspond à celle donnée par fabricant et est en accord avec les taux généralement admis dans la littérature. Pour la spécificité, le principe est la capacité à discriminer les micro-organismes effectivement présents de facteurs interférents, pouvant conduire à de fausses identifications. Ces facteurs peuvent être des mélanges de micro-organismes conduisant à l’identification de micro-organismes qui ne sont en réalité pas présents dans l’échantillon. Des mélanges, des plus éloignés taxonomiquement au plus proches ont été élaborés et analysés (Tableau 4).

5. Résultats et interprétation
Robustesse
Si les études de robustesse sont des requis réglementaires, il s’agit également d’un excellent moyen de mieux caractériser le système pour en faciliter l’usage futur. C’est la conclusion majeure qui a pu être tirée de l’ensemble de cette étude. Pour cela, il est critique d’analyser précisément chaque étape de l’ensemble de la procédure d’identification lors de “l’analyse de risque méthode”. Rapidement lors de l’analyse de risque, beaucoup de questionnements sont apparus sur la partie amont “microbiologie et culture” car le système Maldi Tof est très simple et ne nécessite que très peu de préparation d’échantillon, la qualité initiale de la matière à analyser s’en retrouve donc cruciale. Les recommandations pour effectuer des identifications par Maldi-tof sont généralement : “utiliser une colonie fraiche”. S’il est relativement simple de comprendre cette notion pour un microbiologiste, techniquement lors de l’écriture d’une procédure d’analyse, comment qualifier une “colonie fraiche” ? Moins de 2 jours ? 3 jours ? Est-ce valable sur l’ensemble des différents types de milieux utilisés au laboratoire ? L’appréciation de “fraiche” est-elle identique entre opérateurs ? Une colonie conservée au frigo pendant 3 jours est-elle encore considérée comme fraiche ?…
L’essentiel de l’étude a donc été porté sur cet aspect, un exemple de comparaison de conditions est présenté dans le Tableau 5.
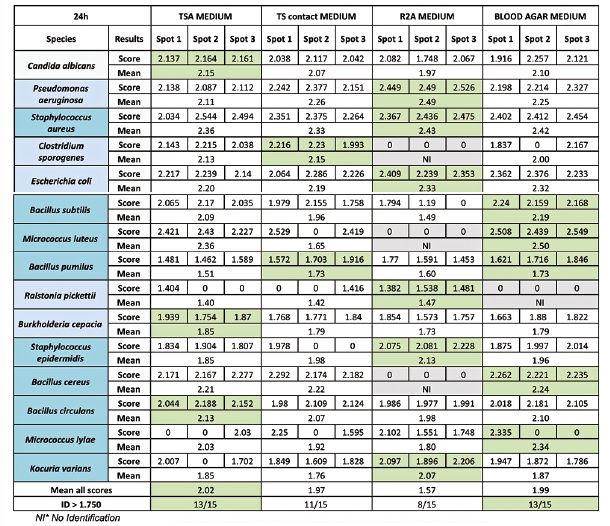
Ci-après les principales conclusions issues de l’étude de robustesse :
- Un milieu gélose au sang ou trypcase soja doit être privilégié,
- Une identification effectuée avant 72 heures garantit les meilleurs résultats,
- Le milieu R2A est trop pauvre pour pouvoir donner une bonne identification rapidement sous 72heures ; les meilleurs résultats sont obtenus sous 5 à 7 jours,
- Les milieux Cétrimide & Mannitol Sel génèrent des résultats similaires à ceux obtenus sur TSA.
- Pour les levures, les identifications peuvent être effectuée jusqu’à 120 heures,
- Un stockage des colonies à froid (+ 2/8°C) pendant 24 heures n’engendre pas d’impact,
- L’étape de dépôt d’acide formique ne doit pas dépasser 30 minutes, il s’agit d’une étape critique,
- Pour la matrice HCCA, le run peut être lancé dès que le dépôt est visuellement sec,
- La réalisation d’au moins 3 tests d’identification sur un dépôt n’a aucun impact sur l’identification.
- Une plaque peut être analysée plusieurs jours après sa préparation. Toutefois, une analyse lancée à 48 heures maximum assure les meilleurs résultats.
Au-delà de la caractérisation et de la connaissance qu’offre une étude de robustesse sur le système, nous nous sommes rapidement aperçus que ces informations allaient être extrêmement précieuses pour l’élaboration de procédures précises ainsi que pour la gestion de déviations associées.
Exactitude & Spécificité
Concernant l’exactitude, pour les quinze souches testées, une identification significative et conforme a été obtenue pour chacune des souches au niveau de l’espace, la précision est donc de 100%. La spécificité est évaluée en utilisant un mélange de micro-organismes, selon la monographie 5.1.6 le syst.me doit identifier une, deux espèces ou aucune. Pour chaque mélange, au moins une ou deux espèces ont été identifiées, ce résultat souligne la performance du Maldi Tof sur ses capacités de discrimination. Compte tenu de la performance des systèmes génétiques que protéolytiques actuels, notamment vis-à-vis des systèmes biochimiques, la probabilité d’identifier une espèce autre par un mélange est aujourd’hui peu probable.
Conclusion
Si la validation d’un système d’identification alternatif requiert un investissement financier et humain, l’étape la plus difficile est peut-être de convaincre son organisation d’investir dans ce projet. De manière classique, vous allez devoir établir un “business case” compilant l’avantage en termes de coûts réactifs, de gains de temps technique… Malgré les nombreux avantages, un système Maldi Tof reste un investissement conséquent, sans parler du coût de qualification, notamment des systèmes informatiques. De notre expérience, si d’un point de vue mathématique et financier, le retour sur investissement peut sembler compliqué à justifier, il faut savoir que les gains les plus importants d’une identification alternative sont clairement les plus difficilement chiffrables, car comment estimer le gain :
- De résoudre une investigation “production” 3, 4 ou 5 jours plus tôt ?
- Libérer un lot commercial plus rapidement ?
- De remplacer l’identification “Gram” par le niveau taxonomique “espèce” sur toutes ses identifications ?
- D’avoir un niveau de confiance proche de 100 % dans ses résultats d’identification ?
- De connaitre ses tendances de contamination plus précisément ?
Après plusieurs mois d’implémentation, ce sont ces avantages qui ont modifié le quotidien au laboratoire de contrôle, bien avant les gains sur les temps standards techniques.
Partager l’article
Alexis BILLARD – STALLERGENES GREER
alexis.billard@stallergenesgreer.com
Glossaire
HCCA Acide 2-cyano-3-(4-hydroxyphényl) acrylique
MALDI TOF Matrix Assisted Laser Desorption/Ionisation – Time Of Flight
MAM Méthode Alternative en Microbiologie
R2A Reasoner’s 2A Agar
UFC Unité formant colonie
Références
Références
- K. Bilecen, G. Yaman, U. Ciftci & Y. Rauf Laleli. 2015. Performances and Reliability of Bruker Microflex LT and VITEK MS MALDI-TOF Mass Spectrometry Systems for the Identification of Clinical Microorganisms. BioMed Research International. Volume 2015, Article ID 516410
- A. Croxatto G. Prod’hom & G. Greub. 2012. Applications of MALDI-TOF mass spectrometry in clinical diagnostic microbiology. FEMS Microbiology Reviews, Volume 36, Issue 2, 1, Pages 380–407
- Q-T Chao, T-F Lee, S-H Teng, L-Y Peng, P-H Chen, et al. 2014 Comparison of the Accuracy of Two Conventional Phenotypic Methods and Two MALDI-TOF MS Systems with That of DNA Sequencing Analysis for Correctly Identifying Clinically Encountered Yeasts. PLoS ONE 9(10)
- W. Gen-fu & L. Xiao-hua. 2007. Characterization of predominant bacteria isolates from clean rooms in a pharmaceutical production unit. J Zhejiang Univ Sci B, 8(9):666-672
- W.A. Hyde. 1998. Origin of Bacteria in the Clean Room and Their Growth Requirements. PDA J Pharm Sci and Tech, 52 154-158
- L. Jimenez. 2007. Microbial Diversity in Pharmaceutical Product Recalls and Environments. PDA J Pharm Sci and Tech, 61 383-399
- F.L.C. Pacheco & T.J.A. Pinto. 2010. The Bacterial Diversity of Pharmaceutical Clean Rooms Analyzed by the Fatty Acid Methyl Ester Technique. PDA J Pharm Sci and Tech, 64 156-166
- T. Sandle. 2011. A Review of Cleanroom Microflora: Types, Trends, and Patterns. PDA J Pharm Sci and Tech, 65 392-403
- T. Sandle. 2013. Automated Microbial Identification: A Comparison of USP and EP Approaches. American Pharmaceutical Review 16(4):56-61
- Sterile Drug Products Produced by Aseptic Processing — Current Good Manufacturing Practice. Guidance for Industry. Oct. 2004
- PDA Technical Report 33 (Revised). Evaluation, Validation and Implementation of Alternative and Rapid Microbiological Methods; Parenteral Drug Association: Bethesda, MD, 2013; 30–33
- General Chapter USP 1223 Validation of alternative microbiological methods, current edition
- General Chapter USP 1113 Microbial characterization, identification, and strain typing, current edition
- Monographie 5.1.6. Méthodes alternatives pour le contrôle de la qualité microbiologique :: 07/2017
Références




